Erfahren Sie mehr über das transformative Potenzial der multiplexen Immundetektion
Die Multiplex-Bildgebung spielt eine entscheidende Rolle bei der Krebsforschung, da sie es Wissenschaftlern ermöglicht, tief in die komplexe Mikroumgebung von Tumoren einzudringen und einzigartige Merkmale und dynamische Verhaltensweisen zu enthüllen. Diese revolutionäre Technik macht verschiedene Zellpopulationen und ihre Interaktionen innerhalb der Tumormikroumgebung gleichzeitig sichtbar und bietet so tiefe Einblicke in das komplizierte Ökosystem von Tumoren. Dies ist unerlässlich, um die Reaktion des Tumors auf therapeutische Eingriffe zu interpretieren, Behandlungsergebnisse vorherzusagen und potenzielle Ziele für immuntherapeutische Strategien zu identifizieren. Die Multiplex-Bildgebung ist ein wichtiges Hilfsmittel bei der laufenden Suche nach Krebsstammzellnischen im Leberzellkarzinom.
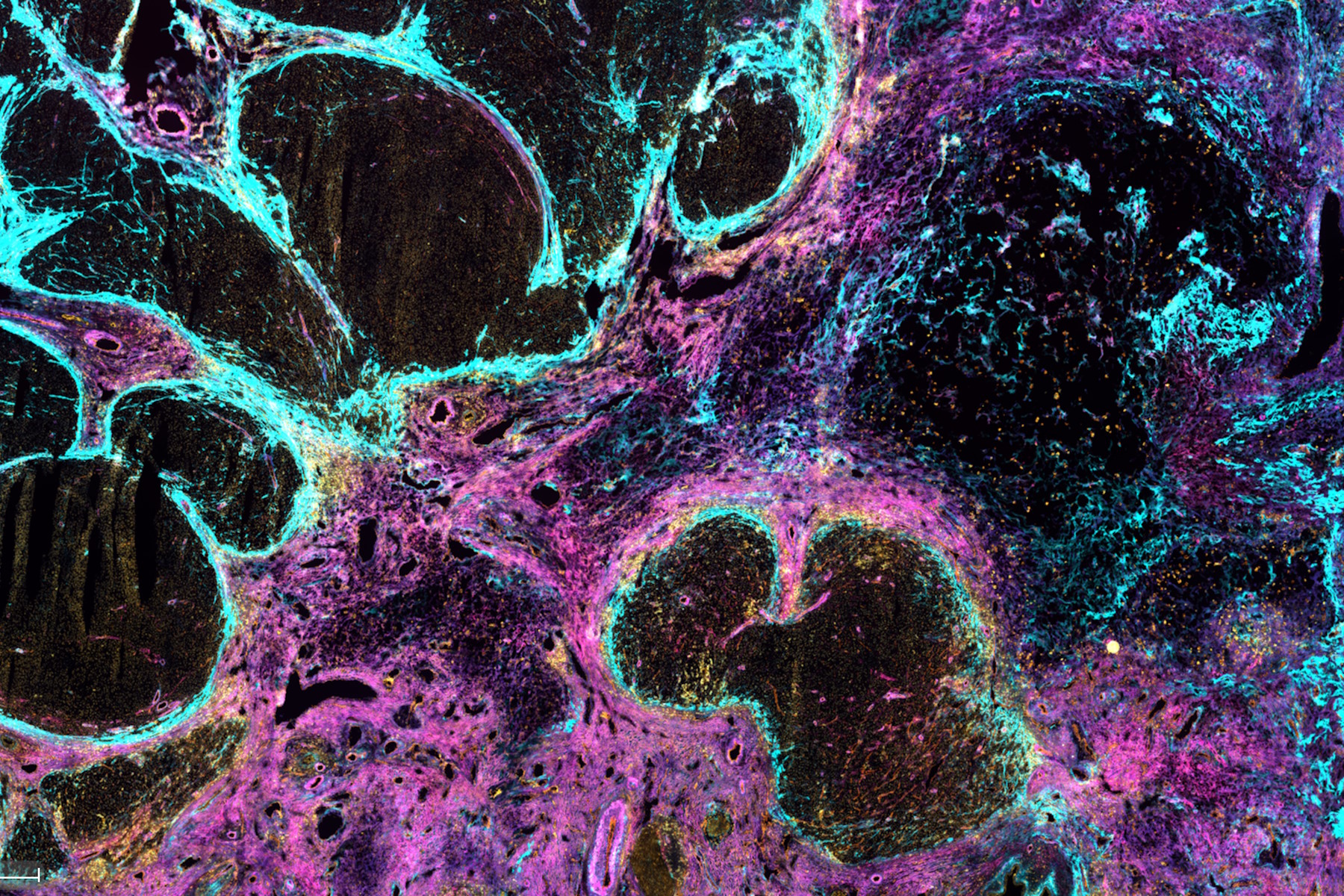
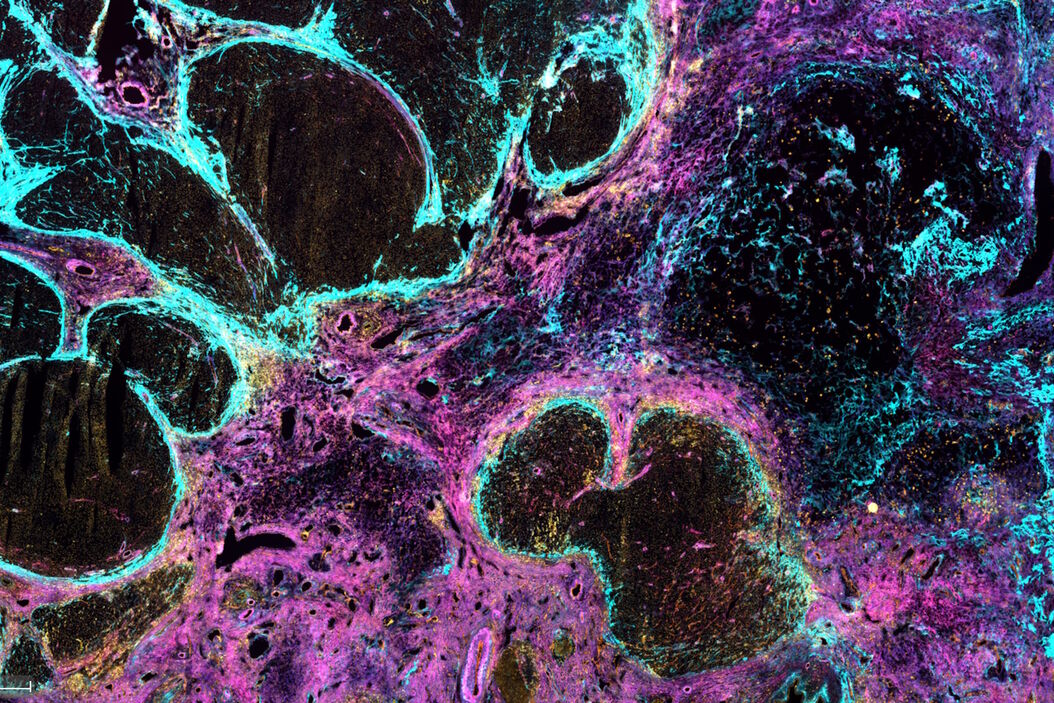
Dieser Anwendungsbericht unterstreicht das transformative Potenzial der Multiplex-Immunodetektion bei der Identifizierung und Charakterisierung von Krebsstammzellnischen im hepatozellulären Karzinom (HCC). Durch die gleichzeitige Erkennung einer Vielzahl von extrazellulären Matrixproteinen (ECM) und Gewebelandmarken zeigt dieser innovative Ansatz die komplexe molekulare Zusammensetzung fibröser Nester auf. Diese spezialisierten Tumor-Mikroumgebungen haben erhebliche Auswirkungen auf das Verständnis des Krankheitsverlaufs und die Überwindung der Behandlungsresistenz.
Die Integration von Proteomik und Multiplex-Bildgebung ermöglicht es Forschern, die komplexen Nuancen von Krebsstammzellnischen beim hepatozellulären Karzinom aufzudecken. Die Fähigkeiten der Cell DIVE Technologie in Verbindung mit dem ClickWell Objektträger vereinfachen den Prozess der Multiplex-Immunfärbung und ermöglichen Einblicke in intratumorale Strukturen, fibröse Nester und die ECM. Mit einer umfassenden Antikörpervalidierung und einem iterativen Färbeansatz, der durch Cell DIVE erleichtert wird, können Forscher zahlreiche Proteine gleichzeitig nachweisen und so ein tieferes Verständnis der Mikroumgebung des Tumors gewinnen.